Product Articles
Life Science
16s Metagenome sequencing และการประยุกต์ใช้กับงานวิจัยด้านต่างๆ

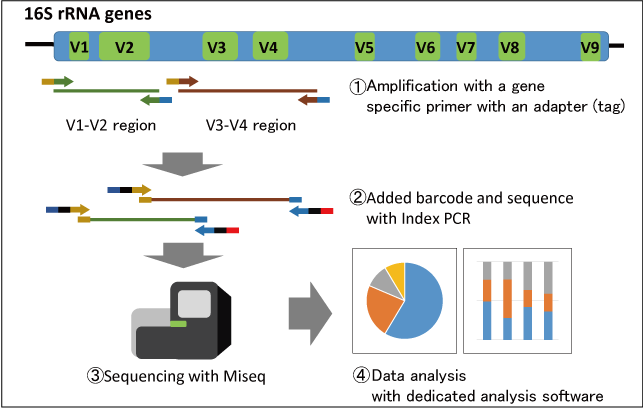
16s Metagenome sequencing เป็นการ sequence ตำแหน่ง 16s rRNA ที่ประกอบด้วย V1-V9 regions เนื่องจากเป็นบริเวณ conserve sequence โดยบริเวณที่นิยมใช้ในการ sequence คือ บริเวณ V3-V4 ที่มีความหลากหลายสูง และจากการศึกษา 16s Metagenome sequencing จะวิเคราะห์ผล Operational Taxonomic Unit (OTU) ที่ทำให้ทราบความหลากหลายของชนิดและปริมาณของแบคทีเรียในตัวอย่าง เพื่อศึกษาความสัมพันธ์ของของแบคทีเรียในรูปแบบต่างๆ อีกทั้งยังสามารถตรวจสอบแบคทีเรียที่ไม่สามารถเพาะเลี้ยงในห้องปฏิบัติการได้ ซึ่งข้อมูลต่าง ๆ เหล่านี้สามารถนำมาประยุกต์ใช้ให้เกิดประโยชน์ในการพัฒนางานด้านต่าง ๆ ที่หลากหลาย เช่น
1. ด้านการแพทย์
2. ด้านสิ่งแวดล้อม
3. ด้านอุตสาหกรรมอาหาร
ขั้นตอนสำหรับการศึกษา 16s Metagenome sequencing ประกอบไปด้วย Sample preparation, Library construction, Sequencing และ Data analysis (Bioinformatics)
ตัวอย่างการประยุกต์ใช้ด้านการแพทย์
- การศึกษา 16s Metagenome sequencing จากตัวอย่างอุจาระผู้ป่วยที่เป็นโรคท้องเสียจำนวน 20 คนในรูปที่ 1
.png)
การศึกษานี้ทำให้ผู้วิจัยพบแบคทีเรียกลุ่มหลักที่เกี่ยวข้องกับผู้ป่วยที่เป็นโรคท้องเสียโดยพบแบคทีเรียในไฟลัม Firmicutes, Bacteroidetes, Proteobacteria และ Actinobacteria จากตัวอย่างทั้ง 20 ตัวอย่าง อีกทั้งพบว่าผู้ป่วยเหล่านี้มีเชื้อโรค Vibrio cholerae และ Helicobacter pylori ที่ยังไม่แสดงอาการของโรค นอกจากนี้ยังทำให้เข้าใจเกี่ยวกับความสัมพันธ์ของ gut microbiome ของผู้ป่วยที่เป็นโรคท้องเสีย
- การศึกษา 16s Metagenome sequencing จากตัวอย่างอุจาระผู้ป่วยจำนวน 3 คน โดยการเก็บตัวอย่างอุจาระในสารต่างชนิดกัน เพื่อศึกษาปัจจัยที่ช่วยในการรักษาสภาพตัวอย่าง โดยทำการเก็บตัวอย่างอุจาระแบบสด, เก็บตัวอย่างอุจาระใน ethanol และตัวอย่างอุจาระใน RNAlater เพื่อที่จะเลือก condition ที่ดีที่สุดในการเก็บตัวอย่าง ในรูปที่ 2
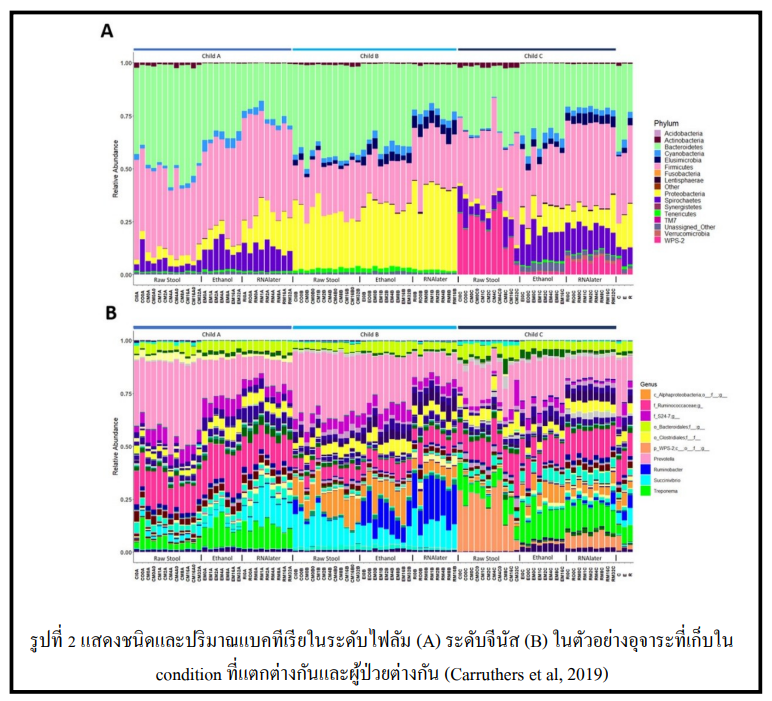
จากการศึกษา 16s Metagenome sequencing ผลการทดลองพบว่าการเก็บตัวอย่างใน condition ที่แตกต่างกันไม่ส่งผลต่อปริมาณแบคทีเรียที่พบอย่างมีนัยสำคัญ
ตัวอย่างการประยุกต์ใช้ด้านสิ่งแวดล้อม
- การศึกษาความหลากหลายของแบคทีเรียในตัวอย่างน้ำพุร้อน โดยการทำ 16s Metagenome sequencing ในรูปที่ 3
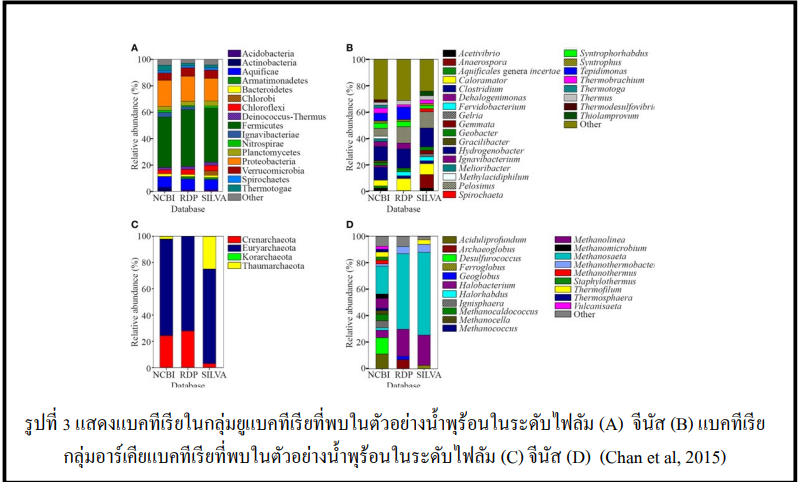
ซึ่งผลการศึกษาพบว่าแบคทีเรียส่วนใหญ่ที่พบ คือ Firmicutes และ Proteobacteria และพบว่าประมาณ 70% ของแบคทีเรียที่พบเป็นแบคทีเรียในกลุ่ม thermophiles
ตัวอย่างการประยุกต์ใช้ด้านอุตสาหกรรมอาหาร
- การศึกษาแบคทีเรียในตัวอย่างกิมจิที่ผ่านกระบวนการหมัก 29 วัน ในรูปที่ 4
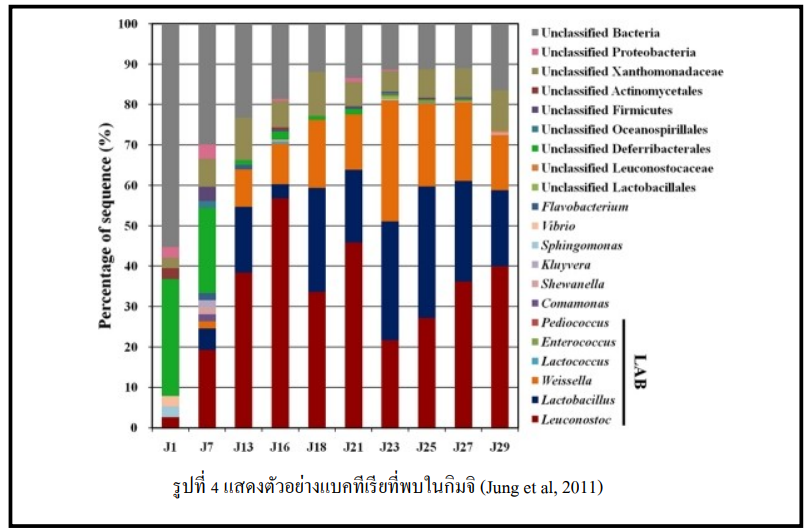
ผลการศึกษาพบว่าแบคทีเรียที่พบในกิมจิส่วนใหญ่ คือ Leuconostoc, Lactobacillus, และ Weissella ซึ่งทำให้เกิดความเข้าใจเกี่ยวกับกลุ่มของแบคทีเรียในตัวอย่างกิมจิ
จากตัวอย่างงานวิจัยดังกล่าวแสดงให้เห็นถึงบทบาทของ 16s Metagenome sequencing ที่สามารถนำไปประยุกต์ใช้ได้กับงานหลากหลายด้าน โดยบริษัทกิบไทยมีบริการ 16s Metagenome sequencing ซึ่งสามารถส่งตัวอย่างได้ทั้งแบบ fresh sample หรือ gDNA extracted โดยเครื่อง sequencer สำหรับ Amplicon metagenome sequencing ที่ใช้คือ Illumina MiSeq / HiSeq2500 platform
สอบถามข้อมูลเพิ่มเติมติดต่อแผนก Technical support e-mail: TAS@3nholding.com
หรือฝ่ายงาน Life Science product โทร 022748331
บทความโดย: กัลยารัตน์ แก้วนิรัตน์
ตำแหน่ง:Technical Application Specialist for NGS and GeneArt Products














