Product Articles
Life Science
เทคนิคการทำ DNA fragmentation สำหรับการเตรียม Library NGS
เทคนิคการทำ DNA fragmentation สำหรับการเตรียม Library NGS
DNA fragmentation เป็นการตัดชิ้นส่วน DNA ออกเป็นชิ้นขนาดเล็กให้อยู่ในช่วงที่ต้องการ โดยทั่วไปจะใช้ชิ้นส่วน DNA ที่มีขนาด 300-600 bp ซึ่งขนาดที่ใช้จะขึ้นอยู่กับรูปแบบการนำไปประยุกต์ใช้ หากนำไปใช้กับ Next-Generation Sequencing (NGS) จะสามารถอ่านลำดับเบสบนสาย DNA เส้นสั้นๆเท่านั้น (short-read sequencing) ซึ่งการทำ fragmentation ในปัจจุบันสามารถทำได้อยู่ 3 วิธี ได้แก่ การแยก DNA เชิงกล (Mechanical Shearing) การย่อยด้วยเอนไซม์ (Enzymatic Digestion) และ การตัด DNA ด้วย Transposon
โดยวิธีที่ได้รับความนิยมคือ Mechanical Shearing เนื่องจากสามารถแยก DNA ได้อย่างอิสระไม่ได้ยึดติดกับ Sequence และได้ขนาดชิ้นส่วนที่สม่ำเสมอมากกว่า (ดังแสดงในภาพที่ 1) ในขณะที่ Enzymatic Digestion ต้องการปริมาณ DNA ที่น้อยกว่าและช่วยให้กระบวนการเตรียมไลบรารีมีความคล่องตัวมากขึ้น
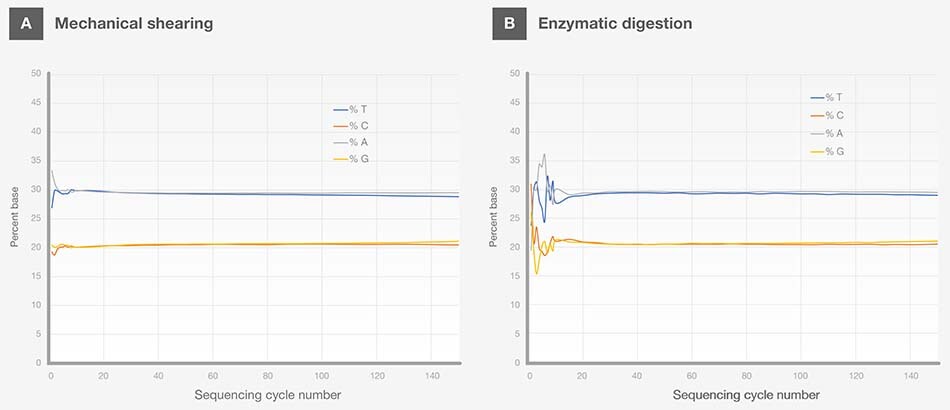
ภาพที่ 1: การเปรียบเทียบเปอร์เซ็นต์ของแต่ละเบสในแต่ละตำแหน่งในการอ่านลำดับเบสระหว่างการเตรียมตัวอย่างด้วยการแยก DNA เชิงกลและการย่อยด้วยเอนไซม์ การแยก DNA เชิงกลแสดงให้เห็นถึงความแปรปรวนเพียงเล็กน้อยในการแทนค่าของเบสที่จุดเริ่มต้นของการอ่านลำดับเบส ขณะที่การย่อยด้วยเอนไซม์มีความแปรปรวนของเบสในช่วงเริ่มต้นของการอ่านลำดับเบสมากกว่าอย่างชัดเจน
1. การแยก DNA เชิงกล (Mechanical Shearing)
การแยก DNA เชิงกลเกิดจาการทำลายพันธะฟอสโฟไดเอสเทอร์ (Phosphodiester Linkages) ด้วยแรงเฉือน (Shearing force) โดยวิธีที่นิยมใช้ในการสร้างแรงเฉือนได้แก่ Sonication, Nebulization, และ Focused Acoustic Shearing
1.1 Sonication
เป็นวิธีที่เรียบง่ายที่สุดในบรรดาวิธีเหล่านี้ โดยใช้เครื่อง Sonicator (ทั้งแบบหัว probe หรือแบบ waterbath) ซึ่งจะทำการปล่อยคลื่นเสียงความถี่ต่ำเพื่อให้ DNA แตกตัว
• แบบหัว Probe (Probe-based Sonication): ปล่อยพลังงานที่มุ่งเน้นไปยังตัวอย่างได้มากกว่า อย่างไรก็ตามตัวอย่างจำเป็นต้องอยู่ในภาชนะเปิดและมีการสัมผัสกับหัว probe โดยตรง ทำให้มีความเสี่ยงต่อการปนเปื้อนสูง
• แบบ Waterbath (Waterbath-based Sonication): ตัวอย่างถูกเก็บไว้ในระบบปิดซึ่งช่วยลดความเสี่ยงในการปนเปื้อน แต่มักจะต้องใช้พลังงานสูงกว่าแบบ probe เนื่องจากการกระจายพลังงานและการสูญเสียพลังงานระหว่างกระบวนการ
อย่างไรก็ตามทั้งสองวิธีจำเป็นต้องมีการปรับแต่งเพื่อให้ได้ความยาวของชิ้นส่วน DNA ตามต้องการ (ดังแสดงในภาพที่ 2) โดยต้องมีการพักตัวอย่างระหว่างรอบการทำ sonicate เพื่อหลีกเลี่ยงความร้อนสะสมซึ่งอาจทำให้ตัวอย่างเกิดความเสียหายและส่งผลทำให้ระยะเวลาในการทำงานยาวนานขึ้น
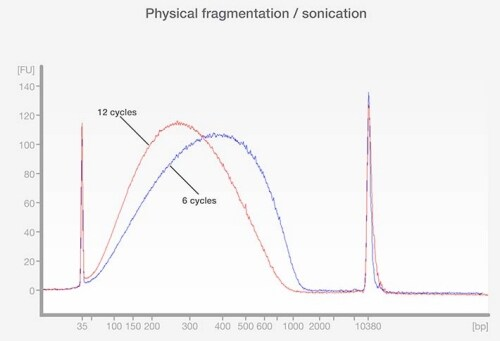
ภาพที่ 2: ความสัมพันธ์ระหว่างการกระจายตัวของค่าความยาวชิ้น DNA กับจำนวนรอบในการทำ sonicate
1.2 Nebulization
วิธีนี้ทำการสร้างแรงเฉือนโดยใช้ก๊าซอัดผ่านรูเล็กๆ ในตัวพ่นละออง (Nebulizer) ทำให้สารละลายที่มี DNA แตกเป็นชิ้นส่วน โดยระดับการแตกตัวสามารถควบคุมได้ด้วยแรงดันของก๊าซ และยังได้รับผลกระทบจากความหนืดและอุณหภูมิของสารละลาย อย่างไรก็ตาม วิธีนี้ต้องใช้ปริมาณตัวอย่างจำนวนมากและมักมีการสูญเสียตัวอย่างจำนวนมาก
1.3 Focused Acoustic Shearing
วิธีนี้ (พัฒนาโดย Covaris) ใช้คลื่นอัลตราโซนิกความถี่สูงเพื่อทำให้ DNA แตกตัว โดยคลื่นความถี่สูงจะทำให้เกิดพลังงานสูงภายในหลอดปิดขนาดเล็ก และยังลดการเกิดความร้อน ด้วยเหตุนี้วิธี Focused Acoustic Shearing จึงได้รับความนิยมในหมู่ผู้ใช้งาน NGS เพราะมีข้อดีเหนือกว่าวิธีดั้งเดิม เช่น
• ลดการสูญเสียตัวอย่าง
• ลดความเสี่ยงต่อการปนเปื้อน
• ควบคุมการแตกตัวให้มีความสม่ำเสมอได้ดีกว่า อย่างไรก็ตามวิธีการนี้จำเป็นต้องมีอุปกรณ์เฉพาะทางและมีต้นทุนที่สูงซึ่งเป็นข้อจำกัดในการใช้งานในบางกรณี
2. การย่อยด้วยเอนไซม์ (Enzymatic Digestion)
การย่อย DNA ด้วยเอนไซม์เป็นทางเลือกที่มีประสิทธิภาพแทนการใช้การตัด DNA ด้วยแรงกล โดยเอนไซม์ที่นิยมใช้ ได้แก่ Endonucleases และ Nicking Enzymes ซึ่งสามารถตัด DNA ได้ทั้งสองสายหรือสร้างรอยนิคในสายเดียวเพื่อให้เกิดการแตกหักของสายคู่ (Double-Stranded Breakage) เพื่อหลีกเลี่ยงการเกิด Sequence Bias จึงมีการใช้เอนไซม์ที่มีความจำเพาะต่ำหรือผสมเอนไซม์หลายชนิดในการย่อย DNA วิธีการนี้มักใช้ DNA ปริมาณน้อยกว่าวิธีการตัด DNA ด้วยแรงกล ทำให้เหมาะสำหรับงานที่มีปริมาณตัวอย่างจำกัด นอกจากนี้การย่อย DNA ด้วยเอนไซม์ยังสามารถดำเนินการเตรียมไลบรารี (Library Preparation) ขั้นถัดไปได้ภายในหลอดเดียวกัน ซึ่งช่วยเพิ่มความสะดวก ลดการสูญเสียตัวอย่าง ลดความเสี่ยงต่อการปนเปื้อน และลดเวลาที่ต้องใช้ในการทำงานด้วย
3. การตัด DNA ด้วย Transposon (Transposon-Based Fragmentation)
อีกทางเลือกหนึ่งที่ผู้ใช้งานบางรายคือการเตรียม Library โดยอาศัย Transposon เป็นวิธีที่แตกต่างจากการตัด DNA ด้วยแรงกลและการย่อยด้วยเอนไซม์ (ดังแสดงในภาพที่ 3) โดยวิธีนี้ใช้ Transposon ในการตัด DNA และในขณะเดียวกันก็ใส่ลำดับ Transposon เข้าไปที่ปลายทั้งสองด้านของชิ้นส่วน DNA ที่ได้ ซึ่งเป็นชิ้นส่วนแบบ Blunt DNA ที่มีลำดับ Transposon ติดอยู่ที่ปลายทั้งสองด้าน จากนั้นจึงเพิ่มอะแดปเตอร์ (Adapters) และ Indexes โดยกระบวนการ PCR ซึ่งวิธีนี้ช่วยลดขั้นตอนดั้งเดิมหลายอย่าง เช่น การตัด DNA แบบดั้งเดิม การแปลงปลาย DNA (End Conversion) และการเชื่อมต่ออะแดปเตอร์ (Adapter Ligation) ทำให้กระบวนการโดยรวมง่ายขึ้นและมีประสิทธิภาพมากขึ้น
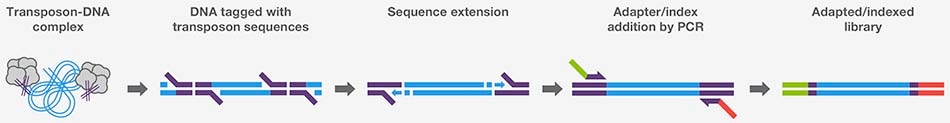
ภาพที่ 3: ขั้นตอนการตัด DNA และเตรียม Library โดยใช้ Transposon
อย่างไรก็ตามการเลือกเทคนิคเพื่อทำ DNA fragmentation สำหรับการเตรียม Library NGS ที่เหมาะสมนั้นขึ้นอยู่กับปัจจัยต่างๆ เช่น ปริมาณตัวอย่างที่มี กระบวนการทำงาน และความแม่นยำของการวิเคราะห์ลำดับเบส
ศึกษาข้อมูลเพิ่มเติม: https://www.thermofisher.com/th/en/home/life-science/cloning/cloning-learning-center/invitrogen-school-of-molecular-biology/next-generation-sequencing/dna-sequencing-preparation-illumina.html
หรือติดต่อแผนก Technical support e-mail: TAS@3nholding.com หรือฝ่ายงาน Life Science product โทร 02-274-8331
บทความโดย: บงกชชนก ด่านรุ่งเจริญ
ตำแหน่ง: Technical Application Specialist














